

8月3日,国际学术期刊Gut在线发表了同济大学生命科学与技术学院生物信息系朱瑞新课题组的最新研究成果“Suppressed hepatic bile acid signalling despite elevated production of primary and secondary bile acids in NAFLD ”。该研究揭示了“肠道菌群通过调控胆汁酸代谢影响FXR信号通路,从而影响非酒精性脂肪肝病(Non-alcoholic fatty liver disease, NAFLD)的病理机制。
NAFLD是与肥胖代谢综合征相关的肝脏疾病,包括单纯性肝脂肪变性(simple liver steatosis)和非酒精性脂肪性肝炎(non-alcoholic steatohepatitis, NASH)。NASH是脂肪变性结合炎症(liver inflammation)和/或纤维化(fibrosis)而形成的NAFLD高级表现。NASH进一步发展可引起更为严重的肝硬化(liver cirrhosis)和肝癌(hepatocellular carcinoma)。肠道菌群(gut microbiota)可以分泌多种酶催化宿主体内的反应,如初级胆汁酸只能在肠道菌群分泌的胆酸水解酶(bile salt hydrolase, BSH)、7-α-脱羟基酶(7-alpha-dehydroxylase)作用下转化为次级胆汁酸。胆汁酸不仅有助于饮食中脂肪的吸收,还能作为信号分子调控肝脏和肠道菌群之间形成的代谢稳态,该过程主要是通过胆汁酸受体farnesoid X receptor (FXR)和G-protein coupled bile acid receptor 1 (GPBAR1 or TGR5)介导的信号进行调控的。然而,之前对于NAFLD病人的研究报道了不一致的胆汁酸水平以及FXR信号变化,肠道菌群参与的胆汁酸调节及其对于NAFLD的影响尚不明晰。
为了明确NAFLD病人中胆汁酸和FXR信号的变化,进一步探究肠道菌群对其调控机制,本研究结合生化检测、转录组学和宏基因组数据,发现NAFLD病人血清中初级胆汁酸和次级胆汁酸绝对浓度增加,其中次级胆汁酸DCA(FXR拮抗剂)的相对含量显著增加,而初级胆汁酸CDCA(FXR激动剂)的相对含量显著降低;同时在NAFLD病人中检测到CYP7A1(rate-limiting enzyme in bile acid synthesis), NTCP和PON1的表达量显著上调,SHP和BSEP的表达量没有显著变化,血清中FGF19含量显著降低,表明NAFLD病人中FXR-medicated和FGFR4-medicated的信号受到压制;此外,NAFLD病人肠道中参与牛磺酸和甘氨酸代谢的细菌丰度显著增加,解释了次级胆汁酸DCA产量增多。增加的DCA含量可以在一定程度上解释被抑制的FXR-medicated和FGFR4-medicated信号。本研究在high-fat diet-fat大鼠中也观测到类似的基因表达和肠道菌群变化。该研究结果表明可以通过靶向调控FXR信号介入NAFLD的治疗,如干预肠道菌群中胆汁酸转化细菌的丰度,为NAFLD的治疗提供了新思路。
朱瑞新课题组博士一年级学生焦娜为第一作者。同济大学生命科学与技术学院朱瑞新博士、纽约州立大学布法罗分校消化道疾病与营养中心Lixin Zhu博士和Susan S. Baker博士是共同通讯作者。
同济大学生物信息系朱瑞新课题组运用信息学方法致力于两个主要研究方向:
1、宏基因组分析方法的发展及其在人类活动等领域的应用:
(1) 寻找真核生物的古菌祖先:综合利用宏基因组数据分析、进化分析和生化分析探讨真核生物细胞功能的进化动力和过程。
(2)“关键菌种(Keystone species)”确定:国际上目前尚无成熟可靠的方法,是与菌群(微生物)相关的环境修复和疾病治疗的瓶颈问题。
2、重大疾病心血管和脂肪肝的病理研究及药物开发:
(1) 阐述心血管和脂肪肝两类主要疾病发病机制:结合高通量组学分析和分子生物学验证,从Host和Microbiome及两者相互作用三个层面系统研究心血管和脂肪肝的发病机制,为后续药物研发确定靶标。
(2) 从深海代谢产物和中药资源中开发相关的治疗药物:充分挖掘深海代谢产物和中药资源,综合运用虚拟筛选、有机合成和药理测试开发心血管和脂肪肝治疗和预防药物或保健品。
更多信息请访问课题组网站: http://cadd.tongji.edu.cn/
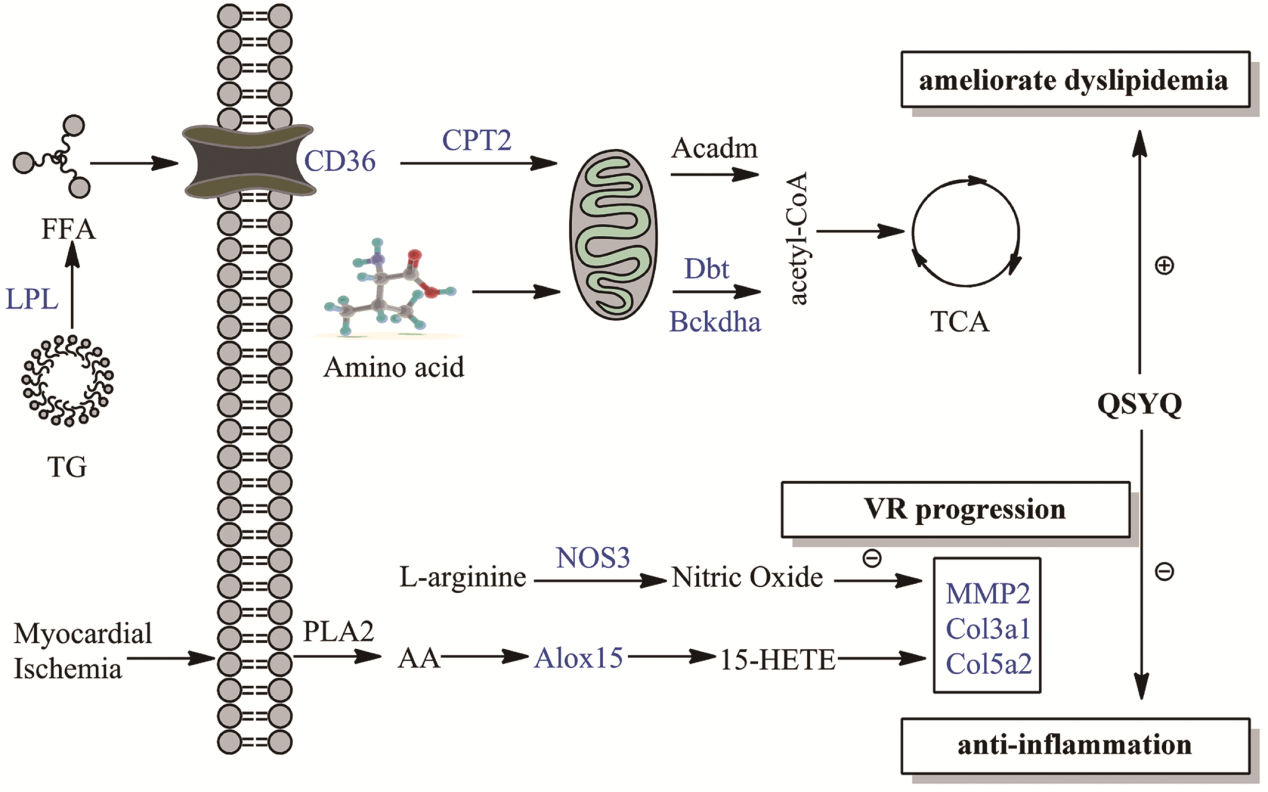
本实验室通过与北京中医药大学王伟教授课题组合作,利用高通量测序技术和生物信息分析对中药复方“芪参颗粒”治疗冠心病心力衰竭的机制进行了详细的研究。发现“芪参颗粒”同时具有“减缓心室重构”、“减弱花生四烯酸脂肪氧合酶代谢和NO产生引起的炎症”和“改善血脂异常”多重能力。该研究不仅同时对中药多个多通路进行了研究,还创新地提出了“基于关键基因的功能分组和重要性评估”方法,为中药多通路机制研究提供通用的生物信息分析方法。
北京中医药大学王勇教授和本实验是林为丽同学为本文并列第一作者,林为丽同学时为同济大学生物信息系2011级本科生。该生基础知识扎实,技术能力全面,异常刻苦,一直是本实验室的骨干成员。林为丽同学承担着本实验室整合组学方向的发展,尤其是本实验的中医药研究方面研究做出了决定性的贡献。祝贺她!
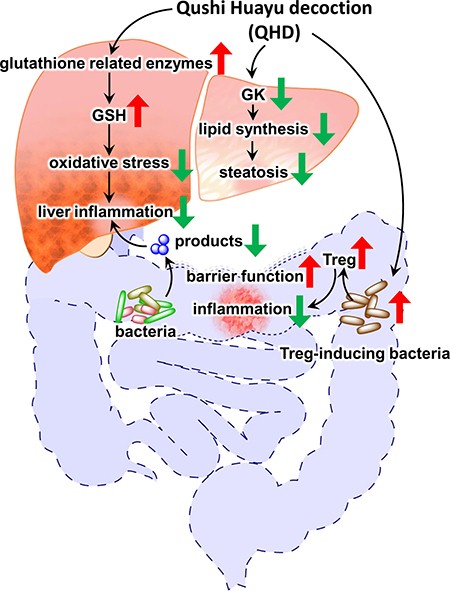
上海中医药大学肝病所胡义扬教授和纽约州立大学布法罗分校,消化道疾病与营养中心实验室,上海中医药大学脾胃病研究所朱立新等人综合运用芯片和宏基因组测序技术对“祛湿化瘀方”(茵陈、栀子、姜黄、虎杖、田基黄)及其中活性成分京尼平苷与绿原酸配伍的“GC方”治疗NAFLD机制进行了研究。发现“祛湿化瘀方”同时具有“增强肝脏的抗氧化”、“降低肝脏脂质合成”和“促进Treg细胞增强肠道屏障”能力。该研究不仅同时对中药多个多通路进行了研究,还为从Host-Microbiome相互作用角度对中药复方的多通路机制研究提供了案例。
本实验室关玲玉和唐诗婕同学参与了本研究的生物信息分析工作。
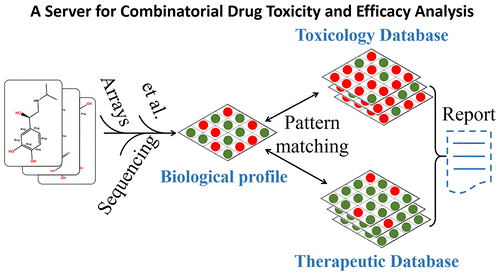
“早发现、早淘汰(Earlier Detection, Earlier Elimination)”对如此耗时耗钱的药物开发尤为重要,但是目前很少有工具能同时提供该服务。为此,我们通过对毒性基因组学和疗效基因组学数据进行无缝整合,开发了第一个能够同时进行先导化合物毒性和疗效预测的在线平台,http://cadd.tongji.edu.cn/webserver/CMCbp.jsp。并且在该平台的开发过程中,我们注重了关键步骤的开放性。也即我们的平台可以非常轻易地拓展到新的比对算法和其它各种新兴的毒性或疗效数据。欢迎使用!
本工作第一作者刘蕾同学时为同济大学生物信息系2012级本科生。本科期间该生就在撰写程序方面表现出超常的天赋,该天赋除了成为了本项目的关键之外,还是实验室网站诞生的基础。目前刘蕾同学在美国辛辛那提大学生物医学信息学(Biomedical informatics at University of Cincinnati)系继续攻读博士学位。祝贺她!
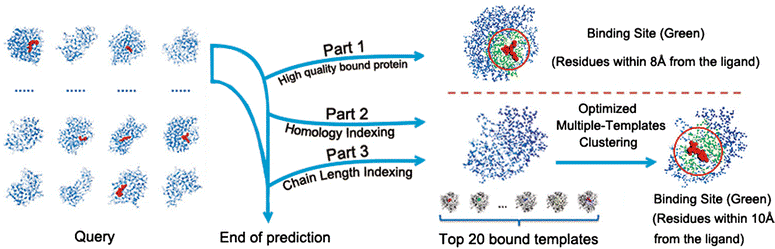
基于受体的药物设计核心步骤首先要进行受体活性位点预测。本研究中,我们充分利用了日益增加的受体-复合物结构信息,经过坐标变化和平移,巧妙地实现了无配体受体的活性位点预测,准确率领跑目前所有已有算法。该算法提供在线服务http://binfo.shmtu.edu.cn/bsitefinder/,欢迎使用!
本工作是本课题组与上海海事大学计算机学院高军博士课题组共同合作完成。
Digestive Disease Week会议是国际消化病领域最权威会议,摘要都需要进行严格peer review。唐诗婕同学现为同济大学生物信息系2012级本科生,是个学习成绩名列前茅,科研效率极高和生活乐趣爆表的现代“三好学生”!再次祝贺她!
Experimental Biology会议是国际上规模最大的综合性生物学大会,摘要都需要进行严格peer review。课题组参与的“Chinese Herbal Medicine for Non-alcoholic Fatty Liver Diseases”工作受邀在EB2016会议作口头报告。课题组唐诗婕同学和关玲玉同学分别参与了该工作的转录组数据和菌群数据分析。这两位同学均为同济大学生物信息系2012级本科生,科研能力突出是她们共同的特点!祝贺她们!
自2012年开始,朱瑞新博士团队对Proteochemometric modeling(PCM)方法及其应用开展了系统的研究。代表性成果包括:1、在对HIV-1蛋白酶多个变体与其抑制剂活性预测研究中,第一次将“蛋白-配体相互作用指纹(protein-ligand interaction fingerprint)”引入了PCM模型中[1]。2、在对HDAC多个亚型与其抑制剂活性预测研究中,创造性地设计了一种新的蛋白描述符用于构建PCM模型[2]。和3、将PCM模型率先用于抗原-抗体相互作用的研究[3]。此外,还对GPCR等最大药物受体开展了PCM方法的应用研究[4]。
在这刚接收的综述中,以自己近年来PCM相关工作为核心,朱瑞新博士团队对PCM模型中的靶点描述符、相互作用项描述符发展以及它们应用进行了系统地综述;最后还对PCM模型可能应用于Target-Catalyst-Ligand系统等研究方向进行了展望。
本综述是在朱瑞新博士指导下,主要由裘天颐同学实施完成。
非酒精性脂肪性肝病(Non-alcoholic fatty liver disease,NAFLD) 包括单纯性肝脂肪变性(simple liver steatosis)和非酒精性脂肪性肝炎(non-alcoholic steatohepatitis, NASH)。 NASH是脂肪变性结合炎症(liver inflammation)和/或纤维化(fibrosis)而形成的NAFLD高级表现。NASH进一步发展可引起更为严重的肝硬化(liver cirrhosis), 和肝癌(hepatocellular carcinoma)。NASH在美国已经成为肝移植第三主因,并预计在2025年跃居成为第一主因。
随着NAFLD患者数量不断增大,NAFLD的研究也在迅速增长,但是目前NAFLD发病机理尚未得到完全阐明。2010年,我们的合作者纽约州立大学布法罗分校,消化道疾病与营养中心实验室主任,上海中医药大学脾胃病研究所执行所长朱立新博士第一次在儿童NASH (pediatric NASH)患者肝组织样本中发现所有乙醇代谢相关通路均上调(mRNA和蛋白水平) [1];2013年进一步在肠道菌群和血液中发现并检测到内源性乙醇,从而提出“肠道内源性乙醇是NASH发病新机制”假说[2]。由于这些工作是在儿童群体中进行,能否在成年人(adult)群体中观察到同样结果是确认这个新假说的一个新挑战。
本工作以前,也有团队对成年人NAFLD患者肝组织的基因表达进行了研究。但是这些工作要么没有与正常肝样本进行比较,要么所使用的“正常肝样本”来源于患有其它慢性肝病、肝硬化或者糖尿病患者。本工作通过严格地选取健康人肝组织样本作为参照,系统地比较了成年人NAFLD患者肝组织基因表达变化情况。在众多显著变化的基因表达和通路中,再次检测到所有乙醇代谢相关通路均上调,从而第一次在成年人群体中验证了“肠道内源性乙醇是NASH发病新机制”假说。
本工作是在朱立新博士和杜克大学医学院胃肠道与肝病中心的Anna Mae Diehl博士共同指导下,多个实验室合作完成。NIH国家过敏和传染病研究所传染病实验室Patrizia Farci博士及其团队为本研究提供了健康人肝组织样本,本实验室林为丽和吴顶峰同学负责了本研究的生物信息分析工作。